Monografías de alumnos: Síndrome de stress porcino o (PSS)
Autora
Garramuño Fernandez, Daiana Elizabeth.
Cátedra:
Genética Básica.
Docentes:
- Prof Asociada: Iglesias, Gabriela.
- Ayudante de Primera: María Pía Beker.
Universidad Nacional de Rio Negro. Carrera de Veterinaria.
Introducción.
Esta monografía tiene como objetivo informar sobre el Síndrome de estrés porcino (PSS) o también conocido como Hipertermia maligna, dando a conocer y detallando características, herencia, frecuencia y métodos de diagnóstico de la misma a partir de recopilación de información de distintos trabajos académicos y artículos.
La mutación.
La presencia de PSS está dada por el alelo recesivo “n” ubicado en el par de cromosomas autosómicos 6, identificándose tres tipos de genotipos posibles: homocigota dominante NN (normal), heterocigota Nn (portador de la mutación) y homocigota recesivo nn (susceptible a la enfermedad).
La mutación se origina de un cambio de citocina por timina en el nucleótido 1843 del gen afectado, esta sustitución provoca un cambio aminoacídico (arginina→ cisteína)[2]en el canal de calcio del retículo sarcoplásmico.
Frecuencia genética.
La frecuencia con que aparece la enfermedad puede variar según las razas y los cruzamientos que se realicen a favor de la economía de la región, siendo más afectada la raza Pietrain con un 97%de los individuos de esta raza, y seguida por 35% en Landrace, 15% en Duroc, 19% en Large White, 14% en Hampshire, 19% en Yorkshire y 16% en razas cruzadas[3].
Diagnóstico.
Existen dos formas de diagnóstico, en la cual una consiste en aplicar Halotano (un gas anestésico), del cual se
origina el nombre Hal para el locus del gen afectado, ya que genera la aparición de síntomas de PSS en aquellos animales que en su genotipo posean el alelo recesivo pero no permite diferenciar los individuos homocigota recesivos que pueden sufrir la enfermedad de los que solo son portadores.
Para esto existe otra forma de diagnóstico: el PCR-RFLP.
La presencia del alelo recesivo genera la aparición adicional de dos secuencias restricción en donde puede actuar una enzima de restricción y cortar el ADN en fragmentos de pares de bases.
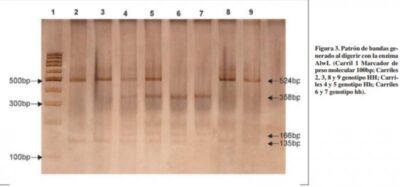
De esta forma los individuos nn presentan los fragmentos 358, 166 y 135 pb, mientras que en los Nn 524, 358, 166 y 135 pb
En el caso de los NN presentan 524 y 135 pb. El fragmento 135 pb es común en los tres genotipos.
El PCR-RFLP (reacción en cadena de polimerasa – polimorfismo en el largo de los fragmentos de restricción) consiste principalmente en replicar muestras de ADN y posteriormente digerirlo con enzimas de restricción cortando cadenas de nucleótidos que permitan la identificación del gen mutado.
Se comienza con el PCR a partir de muestras de ADN (generalmente sangre) a las cuales se las somete procesos de amplificación que incluyen la utilización de dos primers que «flanquean la secuencia de 659pb donde se encuentrala mutación: F-CRC1 5’-TCC AGT TTG CCA CAG GTC CTA CCA-3’ y R-CRC2 3’-ATT CACCGG AGT GGA GTC TCT GAG-5’[4]» (siendo F-CRC1 el cebador iniciador y el R-CRC2 el cebador reverso).
Una vez amplificado el ADN se prosigue con la RFLP en donde se utilizan endonucleasas de restricción para que se unan a secuencias específicas y que a partir de una digestión corten distintos fragmentos del ADN.
En el análisis de PSS se utiliza la endonucleasa la Alw21I (HgiAI) durante 3 h a 37 °C, con una posterior inactivación de la enzima a 65 °C durante 20 minutos y una desnaturalización con proteínasa K, incubándose a 37 °C durante 1 h.[5]
De esta forma se obtiene fragmentos de distintas longitudes que se ven e identifican a partir de una corrida de electroforesis. Para esto se utiliza geles de agarosa y se tiñe la cadena de ADN con bromuro de etidio para poder verlo con luz ultravioleta, el ADN debido al voltaje y el tamaño de la muestra tiende a migrar al polo positivo, pudiendo compararlo con marcadores de peso molecular.
De esta forma se podrán visualizar que fragmentos de secuencias de ADN contiene la muestra.
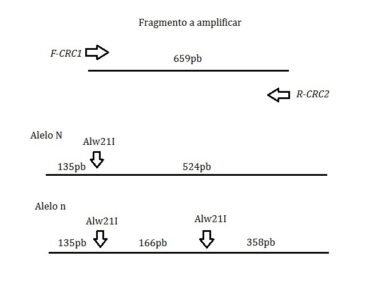
Tamaño del fragmento a amplificar por PCR y un esquema del patrón de restricción con enzimas en el alelo N (dominante) y el n (recesivo) con la enzima Alw211
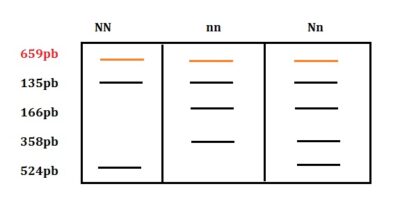
Cómo se verían los genotipos, homocigota dominante a la Izquierda, homocigota recesivo en el centro y Heterocigota a la derecha en geles de agarosa o poliacrilamida
Conclusión.
Es de importancia conocer como reconocer y diagnosticar el PSS con el fin de evitar su propagación y la muerte prematura de animales como también la pérdida de calidad de la carne y sus subproductos.
También es beneficioso como parámetro o tema a considerar para la selección de animales que actualmente debido a la búsqueda de características como aumento de peso y musculatura del animal tienden a propagar este síndrome.
Bibliografía.
- COMA; PIQUER. Avances en nutrición y alimentación animal calidad de carne en porcino: efecto nutrición. Grupo VallCompanys. XV Curso de Especialización .P.8.
- PCR (video obtenido de Roche) y subtítulos en castellano. Canal Youtube Gaby Iglesias.Subido el 6 mar. 2008.
- Técnicas de biología molecular. Blog Desde Mendel hasta las moléculas. Ultima entrada 9/11/2016.
- RIOJAS VALDÉZ; CANALES ZAMBRANO; GÓMEZ DE LA FUENTE; DÁVALOS ARANDA; HERNÁNDEZ VIDAL; SALINAS MELÉNDEZ. Frecuencia alélica del síndrome de estrés porcino en Nuevo León, mediante análisis PCR-RFLP.Facultad de Medicina Veterinaria y Zootecnia, UNAM. Volumen 36 Número 3 Julio-Septiembre 2005. P.2-7.
- MONTENEGRO; CASTRO; BARLOCCO; LLAMBÍ.Frecuencia alélica del Síndrome de Estrés Porcino en Uruguay(análisis por PCR-RFLP).Sociedad de Medicina Veterinaria del Uruguay. Año LX Vol. 46 N° 177-178-179-180 Enero – Diciembre de 2010. P.23-25.
- STALDER; CONATSER. Porcine Stress Syndrome and Its Effects on Maternal, Feedlot and Carcass Quantitative and Qualitative Traits. Agricultural Extension Service The University of Tennessee. PB 1606. P.1-4.
- NONNEMAN; BROWN-BRANDL; JONES; WIEDMANNL; ROHRER. A defect in dystrophin causes a novel porcine stress syndrome.BMC Genomics. USDA, ARS, U.S. Meat Animal Research Center, Clay Center, Nebraska, USA.2012.
- HERNÁNDEZ; POSSO TERRANOVA; MUÑOZ FLÓREZ. Detección de una mutación puntual en el gen receptor de la ryanodina (Ryr 1) en cerdos criollos colombianos. Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, A.A. 237 Palmira, Valle del Cauca, Colombia. 2008. P.3.
[1]COMA; PIQUER. Avances en nutrición y alimentación animal calidad de carne en porcino: efecto nutrición. GrupoVallCompanys.XV Curso de Especialización .P.8.
[2]MONTENEGRO; CASTRO; BARLOCCO; LLAMBÍ. Frecuencia alélica del Síndrome de Estrés Porcino en Uruguay(análisis por PCR-RFLP).Sociedad de Medicina Veterinaria del Uruguay. Año LX Vol. 46 N° 177-178-179-180 Enero – Diciembre de 2010. P.23.
[3]RIOJAS VALDÉZ; CANALES ZAMBRANO; GÓMEZ DE LA FUENTE; DÁVALOS ARANDA; HERNÁNDEZ VIDAL; SALINAS MELÉNDEZ. Frecuencia alélica del síndrome de estrés porcino en Nuevo León, mediante análisisPCR-RFLP.Facultad de Medicina Veterinaria y Zootecnia, UNAM. Volumen 36 Número 3Julio-Septiembre 2005. P.4
[4]MONTENEGRO; CASTRO; BARLOCCO; LLAMBÍ.Frecuencia alélica del Síndrome de Estrés Porcino en Uruguay(análisis por PCR-RFLP).Sociedad de Medicina Veterinaria del Uruguay. Año LX Vol. 46 N° 177-178-179-180 Enero – Diciembre de 2010. P.24.
[5]MONTENEGRO; CASTRO; BARLOCCO; LLAMBÍ. Frecuencia alélica del Síndrome de Estrés Porcino en Uruguay(análisis por PCR-RFLP).Sociedad de Medicina Veterinaria del Uruguay. Año LX Vol. 46 N° 177-178-179-180 Enero – Diciembre de 2010. P.24.