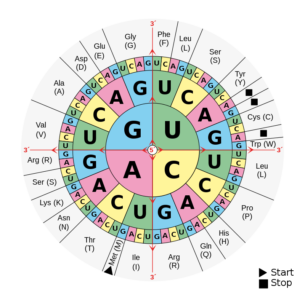
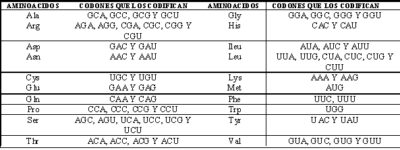
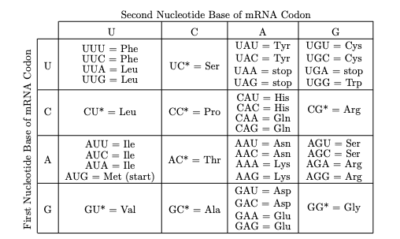
El código del ADN o sea el código genético:
A pesar de que solo hay 20 aminoácidos, Francis Crick descubrió que el código genético se lee de a tres bases.

codigo genético con los aminoácidos y estructura de wikipedia
Obsérvese que la mayoría de los aminoácidos están representados por más de un codón y que la variación entre esos codones para el mismo Aminoácido, en general es en la tercera base.
| Símbolos | Aminoácido | M Wt | Propiedades | Codón (s) | ||
|---|---|---|---|---|---|---|
| Ala | A | Alanina | 89.09 | No esencial | hidrofóbico | GC* |
| Arg | R | Arginina | 174.20 | Esencial | Hidrofílico, básico | CG* AGA AGG |
| Asn | N | Asparagina | 132.12 | No esencial | Hidrofílico, sin carga | AAU AAC |
| Asp | D | Ácido aspártico | 133.10 | No esencial | Hidrofílico, ácido | GAU GAC |
| Cys | C | Cisteina | 121.15 | No esencial | Especial | UGU UGC |
| Gln | Q | Glutamina | 146.15 | No esencial | Hidrofílico, sin carga | CAA CAG |
| Glu | E | Ácido glutámico | 147.13 | No esencial | Hidrofílico, ácido | GAA MORDAZA |
| Gly | G | Glicina | 75.07 | No esencial | Especial | GG* |
| Su | H | Histidina | 155.16 | Esencial | Hidrofílico, sin carga | CAU CAC |
| Ile | I | Isoleucina | 131.17 | Esencial | Hidrofílico, ácido | AUU AUC AUA |
| Leu | L | Leucina | 131.17 | Esencial | Especial | UUA UUG CU* |
| Lys | K | Lisina | 146.19 | Esencial | Hidrofílico, básico | AAA AAG |
| Met | M | Metionina | 149.21 | Esencial | Hidrofóbico | AGO |
| Phe | F | Fenilalanina | 165.19 | Esencial | Hidrofóbico | UUU UUC |
| Pro | P | Prolina | 115.13 | No esencial | Especial | CC* |
| Ser | S | Serina | 105.09 | No esencial | Hidrofílico, sin carga | UC* AGU AGC |
| Thr | T | Treonina | 119.12 | Esencial | Hidrofílico, sin carga | AC* |
| Trp | W | Triptófano | 204.23 | Esencial | Hidrofóbico | UGG |
| Tyr | Y | Tirosina | 181.19 | No esencial | Hidrofóbico | UAU UAC |
| Val | V | Valina | 117.15 | Esencial | Hidrofóbico | GU* |
| iniciar | Metionina | AGO | ||||
| detener | UAA UAG UGA | |||||
Esta sección está basada en notas escritas por Tim Wagner. shown are the one-letter abbreviation for each, its molecular weight, and some of its properties, taken from H. Lodish, D. Baltimore, A. Berk, S. L. Zipursky, P. Matsudaira, and J. Darnell, “Molecular Cell Biology,” third edition, W. H. Freeman and Company, New York, NY; 1995.
El significado de cada uno de los codones se descubrió a través de ensayos in vitro con soluciones de polinucleótidos, ribosomas, una fuente de energía y un mARN; así se descubrió que por ejemplo el ácido poli uridílico codificaba una cadena proteica formada sólo por fenilalanina por lo tanto el codón UUU codifica para este aminoácido.
Así también se encontró que 3 de los 64 codones no codifican para ningún aminoácido (UAG, UGA y UAA) y por lo tanto son codones de fin de cadena o de terminación de la síntesis proteica, también llamados codón Ámbar, Ópalo y Ocre respectivamente.
Por todo esto se descubrió luego que no era necesario que hubiera 64 ARNt para cada triplete sino que existen muchos menos ya que como la tercera base del codón del mensajero es redundante.
La primera base del anticodón puede aparearse de forma no usual aceptando que algunas bases se apareen en forma anormal o se cambia químicamente permitiendo que reconozcan a más de una tercera base. A esto se le llama apareamiento tipo Wobble o de titubeo.
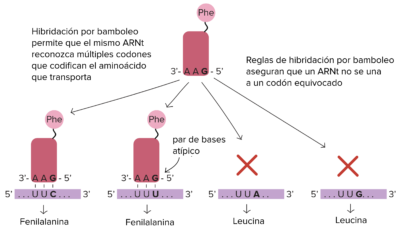
Apareamiento tipo Wooble o apareamiento poco frecuente de los ARN (codón y anticodón). Fuente: Khan Academy
La traducción de proteínas es un proceso rápido en los procariotas casi simultáneo a la transcripción y en los eucariotas en el citoplasma con el ARNm maduro con una proteína que se une a la cola poly A (poly A binding protein) y esto colabora a que la traducción sea más eficiente.
Tiene varios pasos la iniciación en la que difieren las bacterias de los organismos superiores y el resto de las etapas son iguales en ambos organismos. Las siguientes etapas son la elongación y translocación que son seguidas una de otra, dónde comienza a crecer la cadena polipeptídica y la terminación que es el fin de la traducción cuando aparece un codón de stop (UGA; UAA Y UAG).
Fases de la síntesis de proteínas
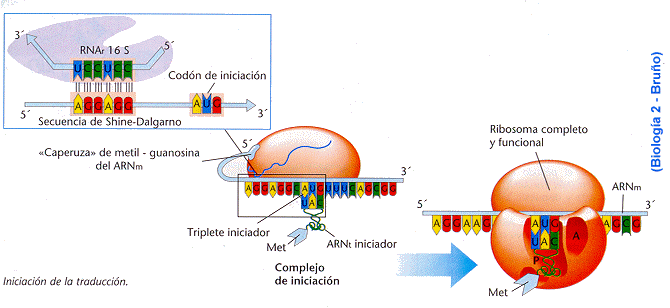
Fase de Iniciación
Cada ARNm tiene una secuencia de bases precedente al codón de inicio que es complementaria a una secuencia de consenso que se encuentra en el ARNr 16 S, la cual se denomina secuencia de Shine Dalgarno y que permite el apareamiento entre los dos ARN (ARNm y ARNr) por complementariedad de bases, formando el complejo de iniciación.
Este complejo está integrado también por proteínas indispensables para el inicio y que son llamadas Factores de iniciación, que intervienen en la disociación de los ribosomas, la unión al ARNm, la unión al ARNt iniciador y alguna otra función desconocida aún. En procariotas se conocen 3 de estos factores, y en eucariotas 11 factores.
El Factor de Iniciación 3 (IF‑3) en procariotas, se une a la subunidad pequeña del ribosoma, y los dos se unen al ARNm, por lo tanto éste participa de la separación de las subunidades del ribosoma, luego el IF‑2 es el encargado de traer al ARNt iniciador (ARNt-f Met). Al unirse el IF‑2 con su ARNt , ambos se asocian con el complejo que se había formado entre la subunidad pequeña, el IF‑3 y el ARNm, y forman entonces en conjunto el Complejo de Iniciación.
Más tarde se une al complejo la subunidad grande del ribosoma, quedando liberados al medio los factores de iniciación.
No se conoce aún con exactitud como el IF‑2 reconoce al ARNt-fMet en los procariotas, pero se supone que sería por tener el grupo amino bloqueado. En los eucariotas en cambio el ARNt lleva metionina normal lo que no lo haría diferente de un ARNt-Met común, pero está comprobado que en su extremo aceptor hay una de las bases que no está unida por puente de Hidrógeno a su complementaria y este cambio de configuración sería lo que lo distingue.
Como se carga cada aminoácido con los ARNt que lo puedan portar?
Carga del aminoácido por la aminoacil sintetaza Fuente: um.es/molecula/dupli03
Es importante aclarar que tanto el ARNt iniciador de procariotas como el de eucariotas tienen propiedades especiales que los distinguen de otros ARNt no iniciadores.
INICIACION DE LA TRADUCCION EN EUCARIOTAS
Secuencia de consenso Kozak
La secuencia descripta por Kozak (1987) se encuentra en la mayoría de los ARNm eucariotas y se describe como 5’ gccRccAUGG 3’, donde R puede ser una purina (A o G) tres bases antes del AUG.
Esta secuencia es reconocida por el ribosoma como sitio de inicio de la traducción. El ribosoma la necesita para saber donde comenzar a traducir. Sería el equivalente a la secuencia de Shine Dalgarno en bacterias.
NO debe confundirse con secuencias denominadas RBS (ribosome binding site) que pueden ser el 5’ cap del mensajero o los sitios internos IRES .
Algunas de las bases son más conservadas que otras y por lo tanto más importantes para que la traducción se produzca. Mayormente las más conservadas son AccAUGG.
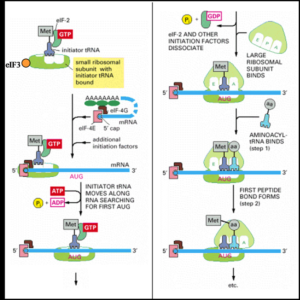
En el caso de los eucariotas hay muchos más factores que intervienen en la iniciación, además de existir algunas diferencias con respecto al ARNt iniciador que porta metionina normal, la subunidad pequeña es 40 S y primero ésta debe reconocer el extremo 5′ del mRNA y desde allí se desplaza sobre el mismo hasta encontrar el codón de inicio (Teoría de scanning). Estas diferencias las realiza gracias a la gran cantidad de factores asociados al ribosoma.
inicio traducción eucariota
Particularidades de la Fase de Iniciación en PROCARIOTAS
El iniciador ARNt-fMet en primera instancia es acilado o cargado con Met, y subsecuentemente modificado o formilado por la ARNt metionil transformilasa que le transfiere un grupo formilo a la Met previamente cargada. Esta enzima transformilasa NO formila la Met de un ARNt-Met no iniciador, por lo tanto debe haber estructuras diferentes entre el ARNt-Met y el ARNt-fMet.
Así también deben existir estructuras que los diferencien en el ribosoma, ya que sólo el ARNt-fMet puede ingresar al sitio P sin ocupar previamente el sitio A como todos los otros ARNt.
Las diferencias encontradas hasta el momento serían por un lado que la base 5′ terminal no tiene puente de H con la base opuesta en el extremo aceptor. Por otro lado existe un puente de H entre un U adyacente al anticodón y una ribosa del anticodón en todos los ARNt que no aparece en el ARNt iniciador, lo que le da una estructura más abierta al loop anticodón. También se describen diferencias en el extremo aceptor y en el loop DHU.
Otra diferencia encontrada es la interacción codón anticodón del ARNt-fMet que podría ser un Wobble en la primera posición del anticodón ya que este ARNt iniciador procariota no sólo puede aparearse con el codón AUG sino también con el codón para Val (GUG) y con los de Leu (UUG y CUG) que ocasionalmente pueden ser usados como codones de paro o stop también.
Este efecto Wobble puede ser explicado por la estructura más abierta del loop anticodón ya mencionada.
Iniciación de la traducción cap dependiente en eucariotas
La iniciación de la traducción en eucariotas involucra ciertas proteínas que tienen una afinidad especial por el 5’ cap propio de los mensajeros eucariotas. Algunas de estas proteínas son la subunidad pequeña 40S del ribosoma y los factores de iniciación que son indispensables para sostener al mensajero en una posición.
El factor eIF3 se asocia con la subunidad pequeña del ribosoma y juega un rol importante manteniendo a las subunidades del mismo separadas evitando su asociación prematura.
A su vez interactúa con el complejo eIF4F que contiene a varios factores: eIF4A, eIF4E and eIF4G. El factor 4E es la proteína de unión al cap o cap binding protein y a veces es clivado por proteínas virales para evitar que la célula infectada traduzca su propios mensajeros.
El eIF4A es una helicasa dependiente de ATP que se encarga de abrir el RNA que se asocia por complementariedad formando horquillas doble hélice.
Otra proteína importante es la Poly-A binding protein o proteína de unión a la cola poly
A de la gran mayoría de los mensajeros. Esta proteína juega un importante rol en la circularización del mensajero durante la traducción.
El complejo de preiniciación formado por la subunidad pequeña y el mensajero acompañados por los factores proteicos realiza el scanning o barrido del mRNA desde el extremo 5’ al 3’. en busca del codón de inicio AUG que codifica para Metionina (Met).
El ARNt iniciador viene cargado con Met formando parte del complejo con el ribosoma y por lo tanto la traducción comenzará con Met, a menudo luego removida por una proteasa.
El ARNt iniciador traído por el factor eIF2, se introduce en el sitio P de la subunidad pequeña del ribosoma y se hidroliza GTP en esa unión. Esto provoca junto con la liberación de algunos factores en la asociación de la subunidad grande (60S). Así el ribosoma una vez completo está listo para iniciar la elongación y translocación hasta encontrar el codón de stop.
Inciación cap independiente.
- El ejemplo más estudiado de la iniciación cap independiente es el de la entrada al sitio interno del Ribosoma (Internal Ribosome Entry Site IRES). En estos casos no es necesario el scannig o barrido. El ribosoma es llevado por factores llamados trans actin (ITAFs derivado de IRES trans actin factors) sorteando el barrido. Este modo de inicio es recientemente conocido y es usado en células que requieren una traducción particular debido a factores de stress o incapacidad de traducir a la mayoría de los mensajeros. Por ejemplo en factores que responden a la apoptosis o respuestas inducidas frente a stress
- Si queres hacer una autoevaluación del tema el link acá o en el siguiente link https://forms.gle/harMa5WR9zGM1L8c8
Quizás te interese descargar el pdf completo del tema para leer online o imprimir o te interesa el libro completo del blog y más aún contenidos de todos los temas de genética clásica y molecular